





公众号:AI科技评论
近日来自 Stanford 的 Anvita Gupta, James Zou 在arXiv 上贴出他们近期的工作,利用 GANs 来生成编码可变长度蛋白质的合成 DNA 序列。
首先需要介绍一下合成生物学。
合成生物学是生物科学在 21 世纪才刚刚出现的一个分支学科,其研究方法就是从最基本的要素系统地去设计和合成生物物质(例如合成蛋白质、DNA 片段等)。近年来,合成生物学成长很快,科学家们已经不局限于非常辛苦地进行基因剪接,而是开始构建遗传密码,以期利用合成的遗传因子构建新的生物体。有人甚至认为合成生物学将催生下一次生物技术革命。合成生物学在很多领域将具有极好的应用前景,例如更有效的疫苗的生产、新药和改进的药物、以生物学为基础的制造、利用可再生能源生产可持续能源、环境污染的生物治理、可以检测有毒化学物质的生物传感器等。

但是,像几乎所有需要借助人工智能的学科一样,目前的合成生物技术大多还是手动,这需要大量的时间、劳力以及丰富的领域经验;另一方面,他们现在有大量的基因组和蛋白质组数据集。于是自然就有人想到是否能够利用 AI 技术,通过揭示这些数据集中的模式,帮助他们设计出最佳的生物分子,从而促进生物分子设计的进程。
生成对抗网络(GANs)则代表了将 AI 技术应用于合成生物学中,来生成真实数据(例如基因、蛋白质、药物等)的一种新颖的方法。作者在本文中即利用了 GANs 技术,生成用来编码可变长度蛋白质的合成 DAN 序列。
当然若要保证合成的分子可以应用于各种真实环境中,则不仅仅是要用 GANs 生成新型的序列,还需要根据所需性质对产生的序列进行优化,例如序列对特定配体的亲和力,或者所生成的大分子的二级结构等。
因此作者在文章中,提出了一种新的利用 GAN 生成 DAN 的反馈循环机制,并使用单独的预测期(称为「函数分析器」)来优化这些序列,以获得期望的属性。
作者使用这个模型做了两个案例实验:1)生成抗菌肽的编码 DAN 序列;2)生成α-螺旋抗菌肽的编码 DAN 序列。其中前者对细菌、病毒和真菌具有广泛的抗菌活性,由于它们通常很短(少于 50 个氨基酸),因此用来作为 GANs 模型的案例很具优势。第二个案例,主要是考虑到蛋白质二级结构(例如α-螺旋或β-折叠)的问题,这种二级机构即使在较短的肽中也会出现。
模型
如下图所示,反馈 GAN 模型(Feedback GAN,FBGAN)由两部分组成。
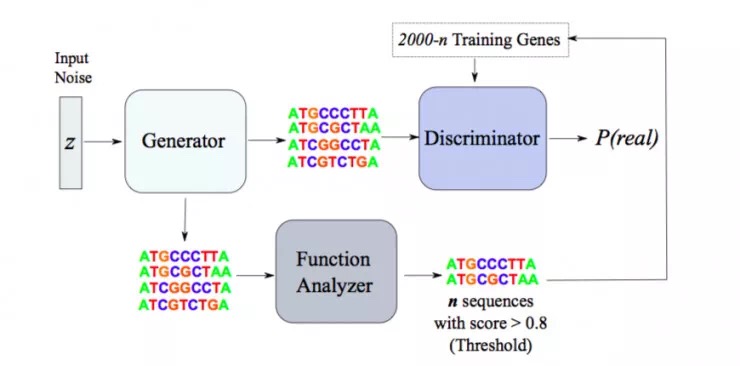
第一个部分为 GAN(准确的说,作者采用了 GAN 的变体 Wasserstein GAN,WGAN),它产生的新型基因序列不具有任何性质。
第二个部分是分析器,在第一个使用案例中,作者选用一个可微分神经网络作为分析器,它接收基因序列并预测序列编码抗菌肽的概率。
事实上分析器是一个黑箱,它的作用就是接收基因序列,并用一个分数来预测基因序列的可取性。例如在α-螺旋肽编码 DAN 序列的案例中,作者用 Web 服务器作为分析器,返回一个基因编码α-螺旋残基的数量。分析器甚至也可以是一个科学家,他们可以通过实验来验证生成的基因序列。
GAN 和分析器在一定的预训练历元(pretraining epochs)后通过反馈机制连接起来,这时候发生器(Generator)才能产生有效序列。一旦反馈机制开始,在每个历元中,发生器 G 产生一定数量的序列,随后输入到分析器中;分析器预测每个基因序列的有利程度,并将 n 个最有利的序列输入到鉴别器(Discriminator)中,作为发生器必须模仿以最小化损失函数的「真实」数据。随后就和通常 GAN 的训练一样了。随着反馈过程的继续,在每个历元中,鉴别器 D 的整个训练集都将被分析器中分数最高的生成序列所替换。
结果
按照上述模型的流程进行试验后,作者通过两项标准测量了 FBGAN 的有效性。
分析器对生成器输出的抗菌性预测是否在不牺牲基因结构的同时随着时间而优化?
从基因序列和所编码的蛋白质性质上来看,产生的基因序列是否与已知抗菌肽基因相似,也即是否过度拟合?
问题一:随时间的优化
为了回答第一个问题,作者检查了在反馈过程中分析器对生成器 G 生成序列的预测情况。如下图所示,经过 10 个闭环训练后,分析器预测大部分序列都是抗菌的;经过 60 个闭环训练后,几乎所有的序列都是高度可能具有抗菌性(大于 0.99)。
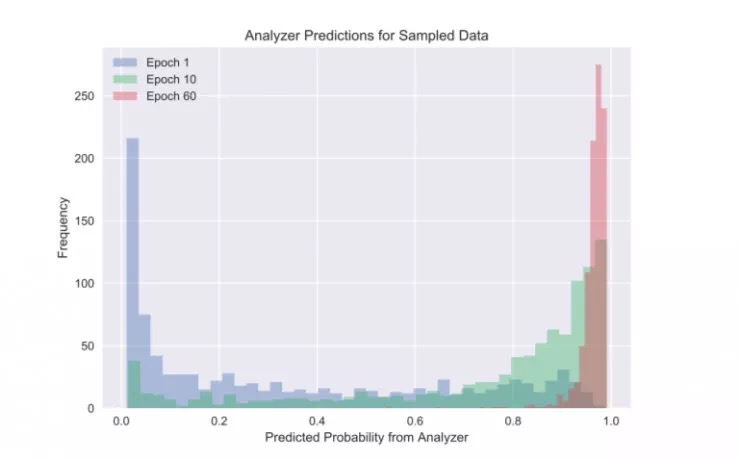
直方图显示了随着闭环训练的进行,产生的基因是抗菌的预测概率。 虽然大多数序列最初被赋予0.1抗菌性,但随着训练的进行,几乎所有的序列最终都被预测为抗微生物,概率大于0.99。

以高于三个阈值 [0.5,0.8,0.99] 的概率预测为抗菌性的序列的百分比。虽然 0.8 被用作反馈的截止点,但在 0.99 以上的序列的百分比在反馈训练期间也继续上升。
值得注意的是,尽管反馈阈值是 0.8,但随着训练的进行预测结果不断提高,甚至远超阈值。这表明闭环训练对阈值的变化是稳健的。此外,闭环训练后产生的序列中 93.3% 的具有正确的基因结构,这表明训练没有牺牲基因结构,反而是被强化了。
问题二:没有过度拟合
如何检测生成序列与实验性抗菌基因的相似性呢?或者说如何判断生成序列没有过拟合呢?这就需要根据编码蛋白质的序列和生理化学性质来判断了。
下图 a 显示了已知抗菌肽和反馈前、后合成基因的蛋白质之间的平均编辑距离直方图。图 b 显示了抗菌肽蛋白内以及反馈后合成基因序列编码的蛋白内的内在编辑距离。所有的编辑距离通过序列的长度进行归一化。从图 a 中,可以看出编辑距离的分布在反馈后向小端发生了移动;而另一方面从图 b 中,反馈后的序列相比抗菌肽序列,有更高的内在编辑距离。这些表明该模型没有过度拟合/复制单个数据点。
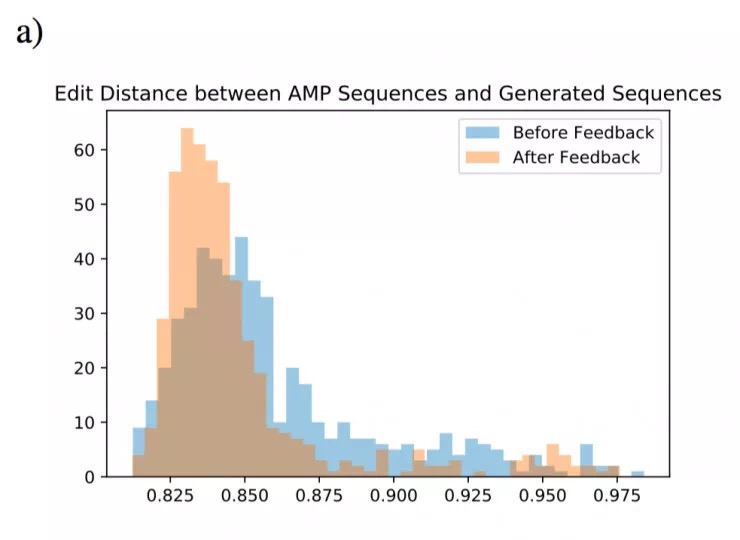
已知抗菌肽序列(AMP)与:1)反馈前产生的合成基因编码的蛋白质;2)反馈后产生的合成基因编码的蛋白质,之间的组间编辑距离(Levenstein distance)。 为了计算组间编辑距离,需要计算每个合成蛋白与每个AMP之间的距离,然后绘制平均值。
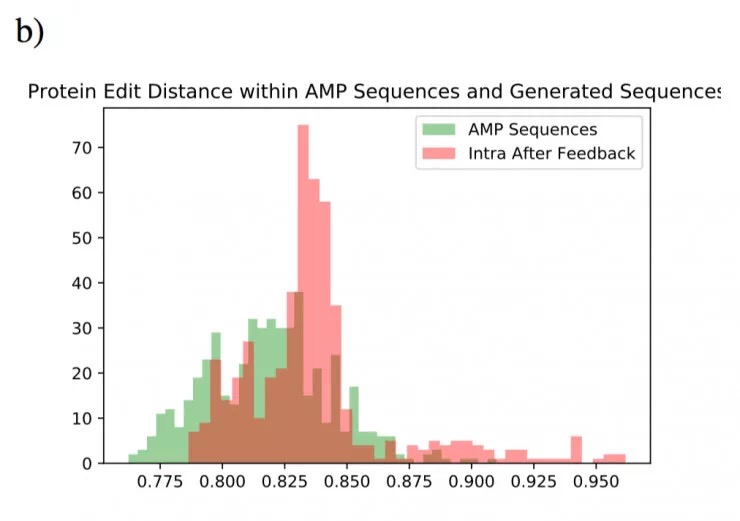
AMPs 和反馈后产生的蛋白质的组内编辑距离,以评估反馈循环后 GAN 产生的基因的变异性。 组内编辑距离通过从组中选择 500 个序列并计算组中每个序列与每个其他序列之间的距离来计算; 然后取这些距离的平均值并绘制出来。
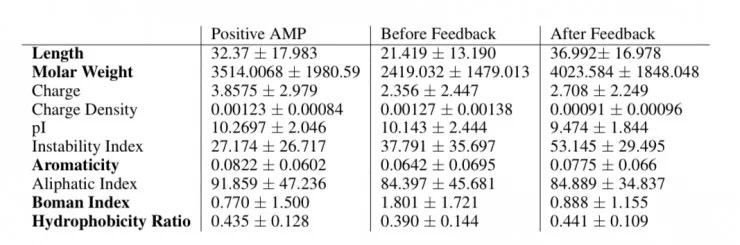
另一方面是通过测量所得蛋白质的生理化学性质来看其相似性,如下表所示。从表中可以看出,由闭环序列编码的蛋白质在十个物理化学性质中有五个(长度、摩尔重量、芳香性、博曼指数、疏水性)在反馈后接近抗菌肽,但其他几个却偏离很大。考虑到分析器只是分析基因序列,而没有考虑这些生理化学性质,所以反馈机制没有直接优化这些性质,也合情合理。
用黑箱 PSIPRED 分析器优化二次结构
用于优化螺旋肽的分析仪是来自 PSIPRED 服务器的黑箱二级结构预测器,它在每个氨基酸处标记具有预测的二级结构的蛋白质序列。所有具有超过 5 个α-螺旋残基的基因序列作为实际数据输入到鉴别器中。
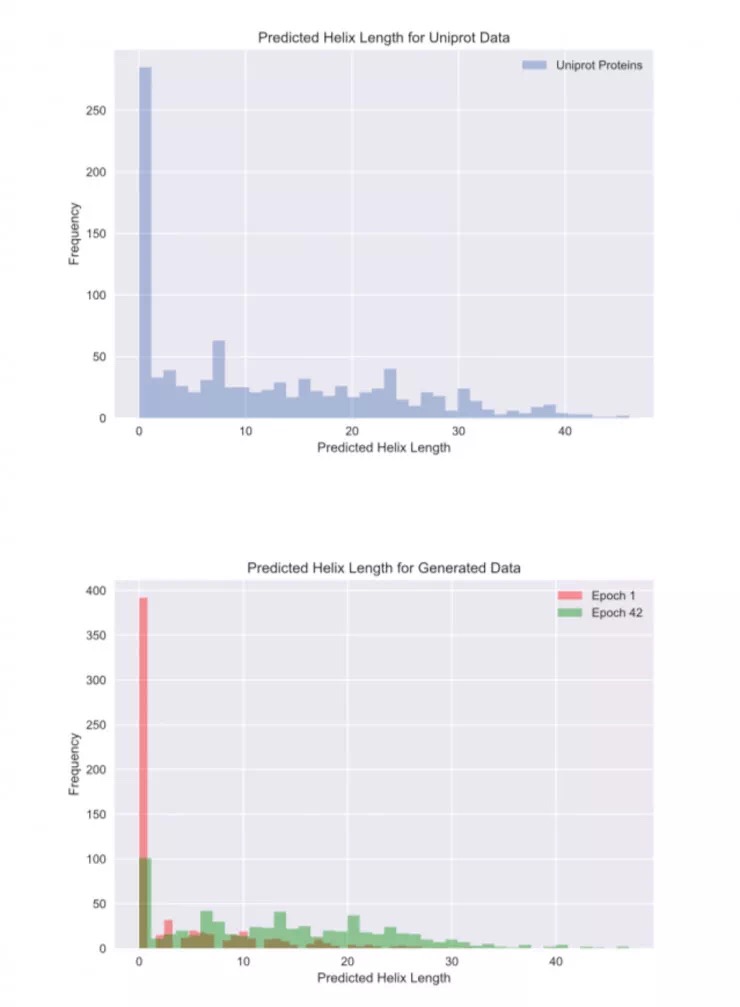
经过 43 次反馈后,生成的序列中的螺旋长度显著高于没有反馈的螺旋长度和原始 Uniprot 蛋白的螺旋长度。
下面为生成的肽的折叠示意图,这两个三维的肽结构是从生成的基因序列中进行从头折叠(ab initio folding)产生的,使用基于知识的力场无模板折叠从 QUARK 服务器。
总结
这个工作的新颖点在于:
首次将 GANs 的技术应用于带有反馈回路机制的生物序列合成;
他们证明了这种训练机制对于所有类型的分析器都有很强的鲁棒性,可以针对特定的特性设计特定的分析器。例如作者分别采用 RNN 分析器和 PSIPRED 分析器优化编码抗菌肽的基因和优化编码α-螺旋肽的基因。
但是这项工作仍然有一些有待改进的地方。例如:
在文中作者限制基因长度为 50 个碱基对,对于较长的基因仍然存在困难,如何将这种方法推广到数千个碱基对的基因序列需要进一步探索;
在文中作者为了降低难度,而专注于生成具有明确的起始/终止密码子结构并且只有四个核苷酸的基因序列,那么能否直接生成蛋白质序列(有 26 个氨基酸)呢?这也需要进一步探索。
论文地址:
https://arxiv.org/abs/1804.01694
论文摘要
生成对抗网络(GANs)代表了一种在合成生物学中产生现实数据(例如基因、蛋白质、药物等)的有吸引力且新颖的方法。在本文中,我们应用 GAN 生成编码可变长度蛋白质的合成 DNA 序列。我们提出了一种新型反馈循环架构,称之为 Feedback GAN(FBGAN)。该模型使用外部函数分析器优化合成基因序列以获得所需特性。我们所提出的这个架构具有分析器不需要可微分的优点。我们将反馈循环机制应用于两个例子:1)产生编码抗菌肽的合成基因;2)优化合成基因用于其所产生肽的二级结构。我们采用几项指标表明 GAN 产生的蛋白质具有理想的生物物理特性。FBGAN 体系结构也可用于优化 GAN 生成的数据点,以获取基因组以外的有用属性。